7 Minuten
Stellen Sie sich einen winzigfliegenden Probenehmer vor, der Blut saugt und dabei unbemerkt die Anwesenheit Dutzender Tierarten in einer Landschaft dokumentiert. Neue Forschungsergebnisse der University of Florida zeigen, dass Mücken-Blutmahlzeiten als kraftvolles, kostengünstiges Instrument für das Biodiversitätsmonitoring dienen können und ein breites Spektrum an Wirbeltieren von Fröschen bis zu Rindern identifizieren.
Wie Mücken zu mobilen eDNA-Probennehmern werden
Ein Forschungsteam unter der Leitung der Entomologen Lawrence Reeves und Hannah Atsma fing über einen Zeitraum von acht Monaten mehr als 50.000 Mücken in einem 10.900 Hektar großen Schutzgebiet in Zentralflorida. Die Fangserie umfasste 21 Mückenarten; aus wenigen tausend blutgefüllten Weibchen extrahierte DNA offenbarte genetische Spuren von 86 verschiedenen Tierarten. Das entspricht ungefähr 80 % der Wirbeltierarten, von denen das Team annahm, dass Mücken in diesem Gebiet Blut saugen würden.
Mittels DNA-Metabarcoding — einer Methode, die kurze, diagnostische DNA-Fragmente amplifiziert und sequenziert — konnten die Forschenden Blutmahlzeiten genetisch bestimmten Arten zuordnen. Das Spektrum reichte über verschiedene Lebensweisen hinweg: baumbewohnende und terrestrische Säugetiere, Amphibien, Zugvögel sowie invasive und bedrohte Arten. Auffällig war das Fehlen bestimmter unterirdisch lebender Tiere wie des östlichen Maulwurfs sowie eines größeren Säugetiers, des bedrohten Florida-Pumas (Puma concolor couguar).
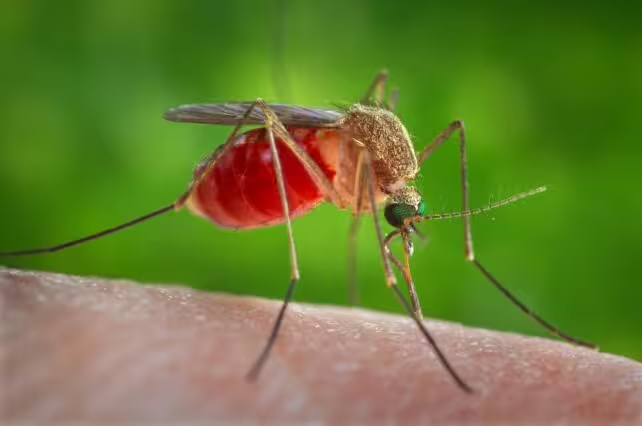
Makroaufnahme einer Mücke mit blutgefülltem Abdomen. Mücken sind biologische DNA-Probennehmer.
Studienaufbau, Methoden und vergleichende Befunde
In einer zweiten ergänzenden Studie unter Leitung des Biologen Sebastian Botero-Cañola verglich das Team die aus Mücken gewonnenen Nachweise mit konventionellen Felduntersuchungen wie Kamerafallen, akustischen Monitorings und visuellen Transekten. Die Probenahme von Mücken während ihrer Aktivitätsphasen erzielte in vielen Fällen vergleichbare Detektionsraten wie direkte Untersuchungsmethoden. In Trockenzeiten hatten traditionelle Methoden jedoch für bestimmte Arten weiterhin Vorteile, etwa wenn Artaktivität saisonal reduziert oder Mückenpopulationen lokal selten waren.
Die Ergebnisse deuten darauf hin, dass die Analyse von Mückenblutmahlzeiten die taxonomische Reichweite von Biodiversitätsbewertungen erweitern kann. Während Kamerafallen oft auf größere Säugetiere fokussiert sind, akustische Detektoren singende Vögel oder rufende Amphibien bevorzugen und visuelle Transekten auf sichtbare Arten beschränkt bleiben, liefert Mücken-eDNA ein gemischtes ‚Schnappschuss‘-Bild: eine Kombination von Arten, die denselben Lebensraum besuchen und von lokalen Mücken gebissen werden. Diese Methode kann so ergänzende Informationen bereitstellen, die klassische Feldmethoden allein nicht liefern.
Warum das für Naturschutz und Monitoring wichtig ist
Felduntersuchungen sind teuer, zeitaufwendig und erfordern oft spezialisiertes taxonomisches Wissen. Mückenbasierte Probenahme reduziert einige dieser Hürden: Fallen sind preiswert, die Feldimplementierung ist relativ unkompliziert, und Laborprotokolle zur Verarbeitung vieler Proben laufen parallelisiert ab. Für ressourcenbegrenzte Naturschutzprogramme kann dieser Ansatz skalierbar sein und helfen, invasive Arten frühzeitig zu erkennen, bedrohte Populationen zu überwachen oder Prioritätsgebiete für intensivere Untersuchungen zu identifizieren.
Reeves merkt zur Ironie an, dass Mücken zwar allgemein verhasst sind, in diesem Kontext aber unbeabsichtigte Wächter der Ökosystemgesundheit darstellen. Atsma und Kolleg:innen betonen jedoch, dass die Technik zwar vielversprechend ist, aber noch Validierung in unterschiedlichen Klimazonen, Ökosystemen und Mückengemeinschaften benötigt, bevor sie als routinemäßiges Überwachungswerkzeug eingesetzt werden kann.
Praktische Grenzen, methodische Feinheiten und nächste Schritte
Es bestehen deutliche Einschränkungen: Spezifische Nahrungspräferenzen der Mücken, jahreszeitliche Schwankungen in ihrer Häufigkeit und die Abbaurate der aufgenommenen DNA beeinflussen alle die Wahrscheinlichkeit eines Nachweises. Die Detektionswahrscheinlichkeit variiert zudem je nach Körpergröße, Verhalten und Häufigkeit der Wirte; häufige, großflächig vorkommende Arten werden statistisch häufiger erfasst als seltene, versteckt lebende oder unterirdische Arten.
Technisch gesehen erfordert die Methode sorgfältige Labor- und Bioinformatik-Workflows, um Fehlzuweisungen und Kontaminationen zu minimieren. Wichtige Schritte umfassen die Auswahl geeigneter DNA-Barcodes/Marker (z. B. COI, 12S, 16S), die Verwendung geeigneter PCR-Primer, Kontrollen für Negativ- und Positivproben, Replikate für die Verifikation sowie stringente Qualitätsfilter in der Sequenzdatenanalyse (z. B. Mindestanzahl an Reads, Abgleich mit zuverlässigen Referenzdatenbanken wie GenBank oder BOLD). Moderne Ansätze wie die Nutzung von Amplicon Sequence Variants (ASVs) statt traditioneller Operational Taxonomic Units (OTUs) erhöhen die Auflösung und Reproduzierbarkeit.
Laborpraktiken beeinflussen die Ergebnisse: Proben sollten rasch konserviert (z. B. in Ethanol oder mittels Trockenmittel) und gekennzeichnet werden, um DNA-Degradation zu minimieren. Zudem sind Protokolle zur Vermeidung von Kreuzkontamination essenziell, z. B. physikalische Trennung von Präparations- und PCR-Bereichen, Einmalwerkzeuge und UV-Decontamination. Bei der Sequenzierung kommen häufig Hochdurchsatzplattformen wie Illumina zum Einsatz, weil sie kosteneffiziente Multiplexierung vieler Proben ermöglichen.
Bioinformatisch sind Entscheidungen über Taxonomiezuordnung und Schwellenwerte kritisch: kurze Barcode-Sequenzen liefern nicht immer eine Auflösung auf Artniveau, insbesondere bei nah verwandten Arten. Ergänzende Daten, etwa lokale Artenlisten, Vorkommensdaten oder historische Inventare, verbessern die Verlässlichkeit der Zuweisungen und reduzieren Fehlinterpretationen. Für robuste Monitoringprogramme sind wiederholte Stichproben und Integration mehrerer Methoden (Kamerafallen, eDNA aus Wasser/ Boden, akustische Überwachung) empfehlenswert, um ein umfassenderes Bild der Artenvielfalt zu erhalten.
Anwendungsbeispiele, Potenzial und Managementimplikationen
Mücken-basierte eDNA-Methoden eröffnen mehrere konkrete Anwendungsszenarien für Forschung und Naturschutz: Frühwarnsysteme für invasive Wirbeltiere (z. B. eingeschleppte Säugetiere oder Amphibien), Überwachung von Weideviehpopulationen in grenznahen Gebieten, Erfassung schwer nachweisbarer Amphibienarten und Dokumentation saisonaler Zugvogelbewegungen. In Schutzgebieten können regelmäßige Mücken-Sampling-Programme als kosteneffiziente Screeningmaßnahme dienen, um Veränderungen in der Artenzusammensetzung zu detektieren und Managementmaßnahmen zeitnah anzupassen.
Für Behörden und Naturschutzorganisationen ist die Skalierbarkeit attraktiv: Durch Kombination standardisierter Feldprotokolle (z. B. definierte Fallenstandorte, gleiche Fangdauer) mit zentralisierten Laborprozessen lassen sich langzeitliche Monitoringreihen aufbauen, die vergleichbare Daten über Raum und Zeit liefern. Solche Daten können in Biodiversitätsdatenbanken integriert werden und als Entscheidungsgrundlage für Schutzmaßnahmen, Beweidungsmanagement oder invasive Artenkontrolle dienen.
Wissenschaftliche Validierung und praktische Empfehlungen
Die Studien in Scientific Reports unterstreichen einen Trend in der Konservationsbiologie: die Nutzung von Organismen und modernen Sequenziermethoden, um Biodiversitätsbefragungen schneller, breiter und günstiger zu machen. Dennoch sollten Anwender folgende praktische Empfehlungen beachten:
- Design und Standortwahl: Fangstellen sollten habitatübergreifend verteilt werden, um unterschiedliche Wirtsgemeinschaften zu erfassen.
- Timing: Probenahme während bekannter Aktivitätsfenster der Mücken verbessert die Trefferquote; mehrere Durchgänge über Saisons geben robustere Ergebnisse.
- Kontrollen: Einbau negativer und positiver Kontrollen in Labor- und Feldprotokolle ist unerlässlich.
- Referenzdatenbank: Aufbau oder Zugang zu lokal validierten Referenzdatenbanken erhöht die Genauigkeit taxonomischer Zuordnungen.
- Hybridansatz: Kombination mit traditionellen Methoden erhöht die Gesamtdetektionswahrscheinlichkeit und kompensiert methodenspezifische Biases.
Langfristig ist es wichtig, protokollierte, standardisierte Methoden zu entwickeln, die verschiedene Regionen und Mückenfaunen berücksichtigen. Interdisziplinäre Zusammenarbeit zwischen Entomologen, Molecular Ecologists, Informatikern und Naturschutzmanagern fördert die Umsetzung in praxisrelevante Monitoringprogramme.
Ethik, Öffentlichkeitsarbeit und Risikobewertung
Obwohl Mücken in der Öffentlichkeit oft nur als lästige Blutsauger oder Krankheitsüberträger wahrgenommen werden, kann die Kommunikation über ihren Nutzeffekt im Naturschutz die Akzeptanz für Feldaktivitäten erhöhen. Transparente Darstellung der Methodik, Datenschutz bei Sensordaten (z. B. bei Kamerafallen) und klare Richtlinien zur Probenlagerung sind Teil guter Praxis. Zudem sollten eventuelle Risiken, wie unbeabsichtigte Verbreitung invasiver Arten durch Forschungsmaterial oder falsche Interpretationen von eDNA-Daten, in Risikobewertungen berücksichtigt werden.
Fazit: Ergänzung, kein Ersatz
Mücken-basierte eDNA-Analysen sind kein Allheilmittel, aber sie erweitern das Werkzeugset für Biodiversitätsüberwachung signifikant. Diese Methode eignet sich besonders als ergänzende Strategie, um Lücken in traditionellen Inventaren zu schließen, frühzeitig Veränderungen zu erkennen und kosteneffizient breitflächige Screeningprogramme umzusetzen. Weitere Validierungsstudien in unterschiedlichen Klimaregionen, gekoppelt mit methodischen Standards, werden entscheidend sein, um das volle Potenzial dieser innovativen Monitoring-Strategie auszuschöpfen.
Beide Studien wurden in Scientific Reports veröffentlicht und betonen eine wichtige Entwicklung in der Naturschutzbiologie: die kombinierte Nutzung lebender Organismen als Probennehmer und moderner Sequenzierverfahren, um Biodiversitätsbefragungen schneller, umfassender und bezahlbarer zu gestalten.
Quelle: sciencealert


Kommentar hinterlassen