8 Minuten
Forscher haben eines der detailliertesten virtuellen Gehirne bisher geschaffen: eine vollständige Simulation des Mauskortex, die einzelne Neuronen und deren Verbindungen nachbildet und neue Experimente zu Hirnwellen, Anfällen und Erkrankungen ohne invasive Eingriffe ermöglicht.
Wie der virtuelle Maus-Kortex aufgebaut wurde
Ein gemeinsames Team des Allen Institute (USA) und der University of Electro-Communications (Japan) nutzte Zellendatenbanken, anatomische Karten und neue, auf Effizienz fokussierte Software, um ein dreidimensionales Modell eines gesamten Mauskortex zusammenzufügen. Der virtuelle Aufbau enthält etwa 9 Millionen Neuronen, verbunden durch rund 26 Milliarden Synapsen, verteilt auf 86 miteinander verknüpfte Regionen. Das Modell läuft auf Japans Supercomputer Fugaku und führt Berechnungen in Quadrillionen-Pro-Sekunde-Skala aus, um realistische neuronale Aktivität zu simulieren.
Zum Vergleich: Ein echtes vollständiges Mausgehirn enthält rund 70 Millionen Neuronen, die in etwa der Größe einer Mandel untergebracht sind. Der simulierte Kortex stellt damit einen Teil des gesamten Organs dar, bewahrt jedoch wesentliche strukturelle und dynamische Details, die biologische Schaltkreise widerspiegeln. Die Forschung zeigt, dass mit ausreichenden Rechenressourcen und präzisen biologischen Daten großmaßstäbliche, biologisch gebundene Gehirnmodelle machbar sind.
Die Entwicklung einer solchen Simulation erfordert die Kombination mehrerer Datendomänen: morphologische Zellprofile, elektrophysiologische Kennwerte, lokale Konnektivitätsmuster sowie makroskopische Verknüpfungen zwischen Regionen. Durch die Harmonisierung dieser Datenquellen lassen sich Modelle erstellen, die nicht nur qualitativ, sondern auch quantitativ konsistente Ergebnisse im Vergleich zu experimentellen Messungen liefern.
Zusätzlich zu den reinen Strukturdaten wurden Wahrscheinlichkeitsmodelle für Synapsenverteilung und Leitfähigkeit implementiert, um Variabilität und Heterogenität innerhalb von Zellpopulationen realistisch abzubilden. Solche Details sind entscheidend, um Phänomene wie heterogene Erregbarkeit, lokale Refraktärzeiten und synaptische Plastizität in Simulationen zu reproduzieren.
Was die Simulation Forschenden erlaubt
Das Modell bietet Forschenden eine dynamische, dreidimensionale Karte, die zeigt, wie einzelne Neurone zeitlich feuern und Netzwerke bilden. Dieses Granularitätsniveau erlaubt es, Hypothesen zur Kognition, zur Ausbreitung von Anfällen, zur interhemisphärischen Kommunikation und zur Entstehung von Hirnwellen in silico zu prüfen — vor oder anstelle kostspieliger bzw. invasiver Tierversuche.
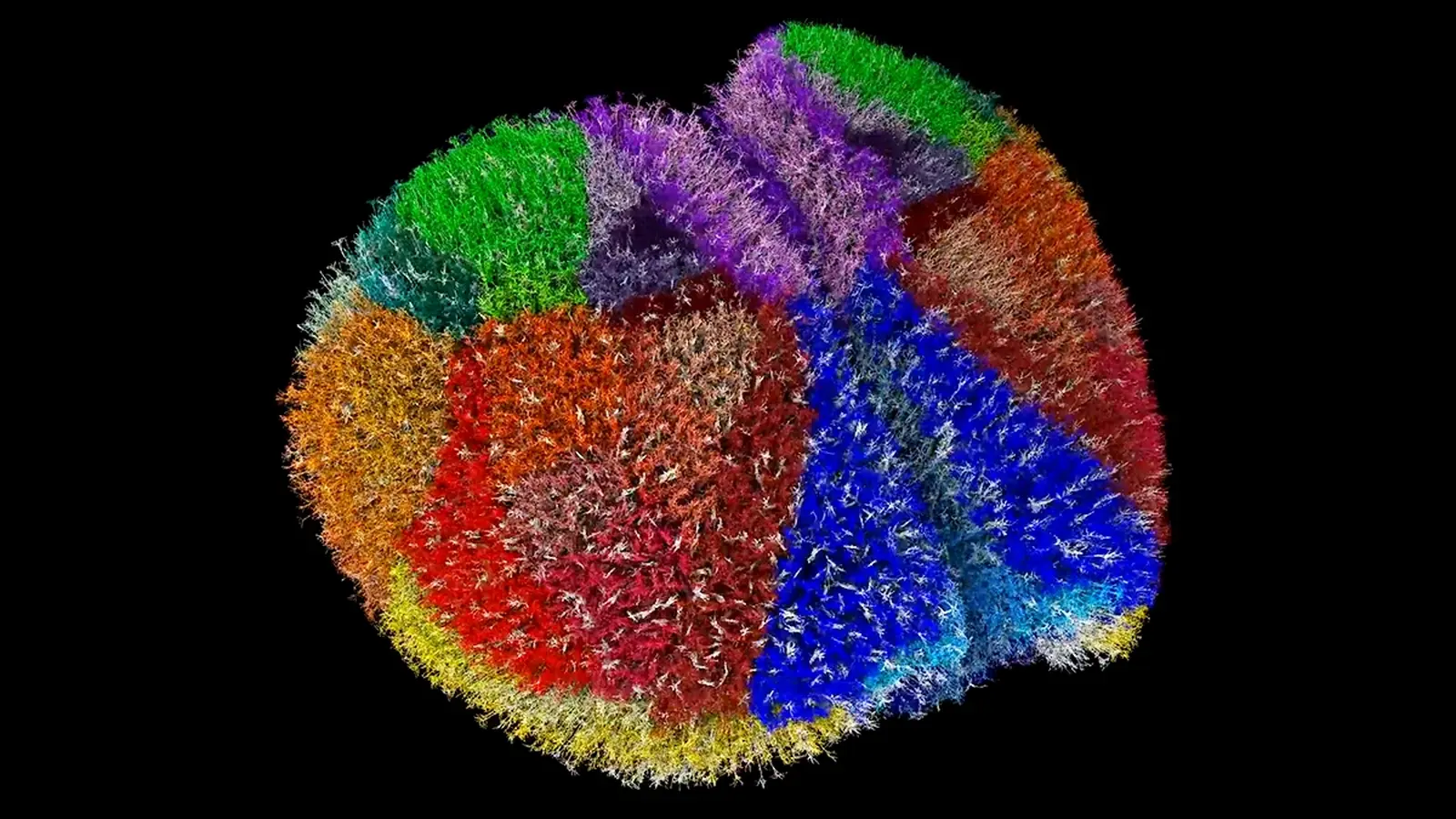
.avif)
Die Simulation ermöglicht es Forschenden, die Aktivität einzelner Neurone zu verfolgen.
Beispielsweise können Teams eine lokale Aktivitätswolke im virtuellen Kortex auslösen, um zu beobachten, wie abnorme Rhythmen sich ausbreiten, oder untersuchen, wie synchronisierte Oszillationen zwischen beiden Hemisphären aufmerksamkeitsähnliche Netzwerkzustände beeinflussen. Da die Software darauf ausgelegt ist, unnötige Rechenoperationen zu minimieren, werden langandauernde Experimente und umfangreiche Parameteruntersuchungen auf heutigen Supercomputern realisierbar.
Die Möglichkeit, detaillierte Stimulationen und Läsionen virtuell durchzuführen, eröffnet neue Wege zur Untersuchung von Ursache-Wirkungs-Beziehungen in neuronalen Schaltkreisen. Forscher können kontrolliert einzelne Zelltypen manipulieren, synaptische Stärken verändern oder modulare Netzwerke temporär deaktivieren, um deren Einfluss auf großräumige Dynamiken zu studieren.
„Dies zeigt, dass die Tür offen ist“, sagt der computational Neuroscientist Anton Arkhipov vom Allen Institute. „Wir können diese Arten von Gehirnsimulationen effektiv betreiben, wenn genügend Rechenleistung vorhanden ist. Es ist ein technischer Meilenstein, der uns Vertrauen gibt, dass deutlich größere Modelle nicht nur möglich, sondern mit Präzision und in großem Maßstab erreichbar sind.“
Solche Simulationen bieten auch eine Plattform für methodische Innovationen: neue Messgrößen für Netzwerkstabilität, algorithmische Ansätze zur Identifikation kritischer Knotenpunkte in Konnektivitätsgraphen und Verfahren zur automatisierten Kalibrierung von Modellen anhand multivariater experimenteller Daten.
Wissenschaftlicher Kontext und Implikationen
Großmaßstäbliche Gehirnsimulationen verorten sich an der Schnittstelle von Neurowissenschaft, Hochleistungsrechnen und Data Science. Sie stützen sich auf detaillierte Atlasdaten zu Zelltypen, Connectome-Karten (die zeigen, wie Neurone verschaltet sind) und elektrophysiologische Messungen, die die Aktivität des Modells einschränken. Sind diese Eingaben präzise, können die simulierten Netzwerke messbare Eigenschaften echten Gewebes reproduzieren — etwa Feuerraten, Synchronisationsmuster und Ausbreitungsgeschwindigkeiten von Wellen.
Klinisch können solche Modelle die Forschung zu neurodegenerativen Erkrankungen wie Alzheimer beschleunigen, indem sie systematische Untersuchungen erlauben, wie zelluläre Veränderungen auf Netzwerkdynamik über die Zeit einwirken. Simulationen können zudem die Zahl von Tierversuchen reduzieren, indem sie vielversprechende Interventionen vorab filtern und mögliche Nebenwirkungen von Therapien auf Netzwerkverhalten vorhersagen.
Die Kombination aus hochauflösenden biologischen Daten und leistungsfähiger Simulationstechnik schafft die Basis für prädiktive Modellierung: Wenn Modelle zuverlässig sind, lassen sich Szenarien durchspielen, die in vivo schwer realisierbar wären — etwa graduelle Synapsenverlust-Profile, diffuse Entzündungsprozesse oder therapeutische Stimulationen mit variablen Parametern.
Tadashi Yamazaki, Informatiker an der University of Electro-Communications, betont Fugakus breitere Rolle: „Fugaku wird in vielen Bereichen der computational science eingesetzt, etwa Astronomie, Meteorologie und Wirkstoffforschung, und trägt zur Lösung gesellschaftlicher Probleme bei. Bei diesem Projekt nutzten wir Fugaku für die Simulation neuronaler Schaltkreise.“
Die Verwendung eines nationalen Supercomputers wie Fugaku erlaubt nicht nur enorme Rechenkapazität, sondern auch die Integration komplexer, parallelisierter Algorithmen für neuronale Simulationen. Dazu zählen skalierbare Integratoren, effiziente Speicherstrategien für Konnektivitätsmatrizen und optimierte Kommunikationsschemata zwischen Rechenknoten.
Perspektiven und Herausforderungen
Das Team hinter dem Modell verwendet es bereits zur Untersuchung von Hirnwellen-Synchronisation und Interaktionen zwischen den kortikalen Hemisphären, hat aber ehrgeizigere Pläne: langfristig sollen Ganzhirnmodelle entstehen, einschließlich simulationsgerechter menschlicher Maßstäbe, die alle verfügbaren biologischen Details integrieren. Dieses Ziel steht jedoch vor mehreren großen Hürden.
- Datenvollständigkeit: Menschliche Gehirne sind wesentlich komplexer als ein Mauskortex; die Gewinnung notwendiger hochauflösender Zelltyp- und Konnektivitätskarten bleibt eine große wissenschaftliche Aufgabe.
- Rechenkosten: Simulationen im menschlichen Maßstab erfordern viele Größenordnungen mehr Rechenleistung und optimierte Algorithmen, um praktisch durchführbar zu bleiben.
- Validierung: Modelle müssen kontinuierlich gegen experimentelle Daten validiert werden, um sicherzustellen, dass simulierte Dynamiken Biologie widerspiegeln und nicht Artefakte von Modellierungsentscheidungen sind.
Trotz dieser Herausforderungen stellt die aktuelle Arbeit einen Proof-of-Concept dar. Durch die Kombination präziser biologischer Datensätze mit führender Supercomputing-Infrastruktur sind Forschende einen wichtigen Schritt in Richtung neuer Werkzeuge zur Untersuchung von Gehirnfunktion und -fehlfunktionen gegangen.
Weitere Herausforderungen ergeben sich aus ethischen und regulatorischen Fragen: Wie dürfen und sollen Ergebnisse aus großmaßstäblichen Simulationen in der translationalen Forschung verwendet werden? Welche Standards für Reproduzierbarkeit, Datenfreigabe und Modelltransparenz sind erforderlich, um Vertrauen in die Vorhersagen dieser Modelle zu schaffen?
Hinzu kommen technische Fragen zur Skalierbarkeit von biophysikalischen Modellen, zur Kompression von Konnektivitätsdaten und zur Integration multimodaler menschlicher Daten wie fMRT, EEG/MEG und Einzelzell-Transkriptomik, um valide, übertragbare Simulationen zu erreichen.
Expertinnen- und Experteneinschätzung
Dr. Maya Fernandez, Neurowissenschaftlerin und Computational Modeler an einem universitären Neurotechnik-Labor, kommentierte die Arbeit: „Großmaßstäbliche kortikale Modelle markieren einen Wendepunkt für die translationale Neurowissenschaft. Sie erlauben es uns, Fragen auf Mechanismus-Ebene zu untersuchen — wie sich zelluläre Pathologien auf Schaltkreis-Funktion ausdehnen — ohne die Störfaktoren lebender Systeme. Der entscheidende nächste Schritt ist die Integration multimodaler humaner Daten, damit Simulationen klinisch relevante Strategien sinnvoll informieren können.“
Solche Statements unterstreichen die Bedeutung von interdisziplinären Kooperationen: Neurowissenschaftler, Informatiker, Data Scientists und Kliniker müssen zusammenarbeiten, um Modelle robust, erklärbar und klinisch nützlich zu machen. Ebenso notwendig sind offene Datenplattformen und standardisierte Formate für Konnektivitäts- und Zelltyp-Informationen, um Vergleichbarkeit zwischen Modellen zu gewährleisten.
Ausblick
Die Simulation wurde auf der SC25 Supercomputing-Konferenz vorgestellt und ist online verfügbar. Sie stellt einen wichtigen Fortschritt in der biologisch fundierten neuronalen Modellierung dar und zeigt, wie Supercomputer wie Fugaku Entdeckungen von der Grundlagenforschung bis zu potenziellen klinischen Anwendungen beschleunigen können. Mit Verbesserungen in Datensätzen und Algorithmen werden virtuelle Gehirnplattformen zu immer mächtigeren Werkzeugen für das Verständnis von Denken, Krankheit und den emergenten Dynamiken neuronalen Gewebes.
Langfristig könnten solche Plattformen in der personalisierten Medizin eine Rolle spielen: patientenspezifische Daten könnten die Grundlage für virtuelle Testläufe von Therapien bilden, um Wirksamkeit und Risiken individuell abzuschätzen. Gleichzeitig bieten Simulationen die Chance, neue Biomarker auf Netzwerkebene zu identifizieren, die in der klinischen Diagnostik und Therapieüberwachung genutzt werden könnten.
Abschließend bleibt festzuhalten, dass diese Arbeiten den Weg für eine neue Generation computationaler Neuroforschung ebnen. Durch die Kombination von detaillierten biologischen Daten, skalierbaren Algorithmen und Hochleistungsrechnern lassen sich komplexe Fragestellungen zur Gehirnfunktion mit einer Präzision untersuchen, die früher undenkbar war. Damit wachsen zugleich die Anforderungen an Datenqualität, Validierung und interdisziplinäre Zusammenarbeit, um das volle Potenzial virtueller Gehirne verantwortungsvoll auszuschöpfen.
Quelle: sciencealert

Kommentar hinterlassen